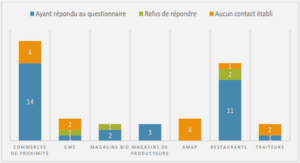
Qu’est-ce que le transcriptome ?
Le transcriptome est un terme générique représentant un ensemble de molécules d’acide ribonucléique (ARN). Ces ARN sont transcrits à partir du génome. Les transcriptomes diffèrent selon le type cellulaire, le moment du recueil et les stimulations reçues par la cellule. Le génome, qui est composé d’acide désoxyribonucléique (ADN), est identique dans l’ensemble des cellules d’un organisme. Par contre, son expression varie au cours du temps. Par conséquent, le transcriptome est une photographie « instantanée » de l’expression du génome. Par extension, le terme transcriptome est utilisé pour les ARN extraits à partir d’un type cellulaire ou d’un tissu.
Le transcriptome regroupe l’ensemble des molécules d’ARN transcrites à partir du génome [15]. Cela comprend les ARN messagers (ARNm), qui sont traduits par les ribosomes en protéines, et les ARN ribosomiques, les ARN de transfert, et tous les autres ARN non codant [16] qui assurent diverses fonctions au sein de la cellule. L’expression du génome met en jeu un grand nombre de mécanismes de modifications post-transcriptionnelles des ARN. Par conséquent, l’ensemble théorique des ARN synthétisés à partir du génome est plus vaste que le génome lui-même. Une des caractéristiques du transcriptome est donc de varier en fonction des conditions et au cours du temps.
Méthodes d’analyse du transcriptome
Il existe de nombreuses méthodes d’analyse du transcriptome : analyse basée sur le type d’ARN étudiés, analyse d’un ou de quelques ARN messagers (par PCR, « Polymerase Chain Reaction » en anglais), ou analyse d’un ensemble d’ARN. Historiquement, la comparaison de deux transcriptomes faisait appel à la construction d’une banque soustractive (figure 4) [17]. Cette méthode est basée sur la propriété d’hybridation complémentaire des acides nucléiques. Elle est qualitative et non exhaustive. Elle identifie un certain nombre de gènes dont l’expression varie entre les deux transcriptomes étudiés (par exemple TA et TB, figure 4). Pour cela, les ARNm sont rétro-transcrits en ADN complémentaires (ADNc) à double-brin. L’un des deux transcriptomes est rétro-transcrit en incorporant des nucléotides marqués (TB, par exemple par de la biotine). Les ADNc des deux transcriptomes sont alors mélangés afin de permettre l’hybridation des ADNc identiques dans les deux transcriptomes. Les ADNc du transcriptome marqué (TB), ainsi que les ADNc hybrides entre les deux transcriptomes sont alors éliminés en utilisant un ligand (streptavidine si les nucléotides sont marqués à la biotine). Il ne reste donc que les ADNc des gènes exprimés uniquement dans le transcriptome A. Ces ADNc sont clonés dans des vecteurs d’expression, puis isolés après transformation dans une bactérie. Les clones d’intérêts sont alors cultivés. Le gène d’intérêt est purifié et séquencé pour identification. Les techniques plus récentes d’analyse du transcriptome permettent une analyse quantitative de l’expression des gènes. La technique SAGE (pour « Serial Analysis of Gene Expression ») est basée sur le séquençage (figure 5) [18]. Les ARNm sont rétrotranscrits en ADNc, puis ces derniers sont soumis à une digestion par un enzyme de restriction. Les fragments obtenus sont ligaturés les uns aux autres et clonés dans un vecteur de séquençage. On obtient des fragments d’ADNc juxtaposés et séparés par un motif connu (le site de l’enzyme de restriction). Le long fragment hybride est séquencé, puis, par analyse bioinformatique, les séquences de chaque fragment sont extraites. Les fragments identiques sont comptabilisés. L’analyse nécessite une étape d’annotation pour identifier le gène dont les fragments sont issus, par alignement sur un génome de référence. Cette technique permet d’obtenir pour chacun des gènes identifiés un compte du nombre de fragments clonés et séquencés. [19]
Les puces à ADN (DNA microarrays) sont un outil d’analyse quantitative de l’expression des gènes (figure 6) permettant l’analyse de l’expression de plusieurs milliers de gènes en une seule expérience [20]. La puce à ADN est généralement une membrane de nylon ou une lame de verre sur laquelle sont attachés des morceaux d’ADN (sondes) dont la séquence est spécifique d’un gène donné. Les sondes sont soit synthétisées in-situ soit déposées par un robot selon un motif régulier et connu. Les sondes sont soit des oligonucléotides (d’une longueur de quelques dizaines de nucléotides), soit des ADNc (d’une longueur de quelques centaines de nucléotides). Les ARNm de l’échantillon étudié sont marqués à l’aide de nucléotides associés à une molécule radioactive ou un fluorochrome au cours d’une étape de rétro-transcription. La sensibilité de l’outil évite normalement une étape d’amplification des ADNc. Selon le même principe d’hybridation spécifique des séquences complémentaires, les ADNc marqués s’hybrident aux sondes complémentaires fixées sur le support. Une fois le support lavé, une image de la puce est réalisée. La fluorescence ou la radioactivité de chaque point de la puce est détectée et convertie en une intensité lumineuse par un scanner approprié. Une analyse bioinformatique permet alors de comparer les quantités relatives de chaque espèce d’ARNm au sein des échantillons. Le séquençage de l’ADN a été mis au point dans les années 1970 indépendamment par Frederick Sanger (Royaume-Uni) et Walter Gilbert (Etats-Unis). Ces deux scientifiques ont reçu en 1980 le prix Nobel de chimie. La méthode de Gilbert ayant recours à des réactifs très toxiques, elle a été progressivement abandonnée au profit de la méthode de Sanger. Cette méthode a connu d’énormes progrès technologiques afin de permettre le séquençage de manière parallèle et à bas coût de génomes eucaryotes entier en peu de temps. La possibilité d’obtenir pour un coût modéré et dans un temps raisonnable les séquences d’un ensemble d’acides nucléiques de grande taille a conduit à transposer cette technique à l’analyse du transcriptome (RNA-seq, figure 7) [21]. La première étape, commune aux autres techniques, est l’extraction de l’ARN. Une des difficultés est d’éliminer les espèces trop abondantes qui risquent de saturer l’analyse, comme par exemple les ARN ribosomiques. Les ARNm sont alors convertis en ADNc. Différents procédés spécifiques [22-27] ont été développés pour retrouver le brin à partir duquel l’ARN a été transcrit, comme par exemple la synthèse d’ADNc mono-brin (Illumina [28]). La phase d’hybridation est virtuelle, par alignement des séquences obtenues avec un génome de référence. Une fois les séquences alignées sur le génome, elles sont dénombrées, ce qui permet une analyse quantitative au niveau de chaque gène.
Réponse à l’infection : données expérimentales
Réponse cellulaire à l’infection
Les puces à ADN permettent (1) d’identifier des signatures moléculaires spécifiques en fonction du stimulus (cellules en culture stimulées par de l’interféron (IFN) α, β ou γ) [30] ; (2) de décrire la réponse de l’hôte à l’infection [31]. En utilisant des cellules dendritiques, impliquées dans l’initiation des réponses immunes innée et acquise, des profils de réponse spécifique et commune à différents micro-organismes ont été caractérisés [32]. Ce résultat a été confirmé par l’analyse de la réponse transcriptionnelle de macrophages humains stimulés ex-vivo par diverses bactéries (E. coli, Salmonella sp., Staphylococcus aureus, Listeria monocytogenes, Mycobacterium tuberculosis, et le bacille de Calmette et Guerrin) [33], et sur sang total, mélangé ex-vivo à du lipopolysaccharide (LPS) ou des fragments de Staphylococcus aureus [34]. La réponse de l’hôte au niveau cellulaire a donc été caractérisée au sein de plus de 25 types cellulaires, après stimulation par des bactéries, virus, levures ou parasites [35]. Ainsi, l’analyse de la réponse transcriptionnelle de l’hôte est un outil de diagnostic moléculaire caractérisant au sein de diverses cellules un profil de réponse temporel précis, sensible et spécifique pour divers micro-organismes.
Spécificité de la réponse (selon le tissu ou le micro-organisme)
Plusieurs équipes ont essayé de confirmer ces résultats in-vivo, dans des modèles animaux de sepsis. L’analyse in-vivo introduit un degré supplémentaire de complexité puisque la réponse de l’hôte peut être observée au sein d’un ou plusieurs tissus. Dans un modèle de sepsis intra-abdominal plurimicrobien (secondaire à une perforation et ligature caecale [CLP]), la réponse tissulaire (analysée au niveau du cerveau, des reins, du foie, des poumons, de la rate et du thymus) était majoritairement spécifique de l’organe, parfois commune à plusieurs organes, ou nettement opposée entre différents organes [36]. L’analyse de la littérature montre que les profils d’expression des gènes diffèrent dans le sang, le tissu hépatique et le tissu splénique [37,38]. La réponse de chaque organe a ainsi été analysée de façon spécifique : cœur [39-42], cerveau [36,43,44], foie [36,37,45-49], rate [36 38,46,47,50,51], poumon [36,47], et sang [38,52,53]. La variabilité des modèles utilisés rend difficile la synthèse de ces résultats. Il apparaît simplement qu’il existe une réponse tissulaire spécifique.
L’utilisation d’un stimulus différent (CLP (polymicrobien) ou LPS) est également à l’origine d’une variation de la réponse [38]. L’analyse du transcriptome aux niveaux sanguin et splénique identifie des signatures spécifiques de chaque stimulus (CLP ou LPS). Dans une autre étude, l’analyse du transcriptome hépatique montre que l’expression de 17 gènes diffère après infection par Escherichia coli ou Staphylococcus aureus [49]. La réponse est donc spécifique, selon l’organe analysé ou la nature de la stimulation.
|
Table des matières
Introduction
Qu’est-ce que le transcriptome ?
Méthodes d’analyse du transcriptome
Réponse à l’infection : données expérimentales
Réponse à l’infection : données humaines
Objectifs
Résultats
Discussion
Conclusion
perspectives
Références bibliographiques
Annexe 1