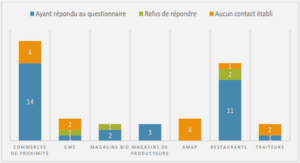
L’implication des laboratoires dans le cadre de la surveillance officielle sur les pathogènes alimentaires Salmonella et Listeria monocytogenes repose sur leur mandat de référence ou sur leur agrément ou reconnaissance délivrés par la DGAL.
En France, pour ce qui concerne les analyses issues de prélèvement humain, la surveillance sur les deux pathogènes est réalisée par le centre national de référence (CNR) de l’Institut Pasteur (Paris). A travers la collecte des souches envoyées par les laboratoires de biologie médicale et hospitaliers, le CNR est en mesure de réaliser une surveillance (évènementielle) des contaminations survenues sur le territoire et de détecter des cas groupés. Le CNR Listeria reçoit également les souches isolées de prélèvements non humains isolées lors d’alertes et d’investigations réalisées sur demande des autorités compétentes pour comparaison génomique avec les souches humaines.
En parallèle, les souches isolées sur la chaîne agro-alimentaires dans le cadre des contrôles programmés (plans de surveillance et de contrôle) par les autorités compétentes, sont collectées et analysées par les laboratoires nationaux de référence (LNR). Un LNR est le garant de la fiabilité des analyses effectuées par l’ensemble des laboratoires agréés et reconnus par l’état. Au niveau Européen, l’ensemble des LNR sont coordonnés par le laboratoire de référence de l’union européenne (LRUE). Celui-ci a pour mission d’harmoniser les pratiques et d’assurer la fiabilité des méthodes d’analyses utilisées par les différents Etats membres.
Au niveau international, un laboratoire peut être désigné comme laboratoire de référence OIE (LR OIE) ou centre de référence FAO (CR FAO). Ainsi, il agit en tant qu’expert sur le pathogène mandaté. L’Agence Nationale de Sécurité Sanitaire de l’alimentation, de l’environnement et du travail (Anses) détient soixante-cinq mandats de référence nationaux, neuf mandats européens et vingt-cinq mandats internationaux.
Les CNR, LNR et LRUE sont chargés, dans le cadre de leurs mandats, d’apporter aux pouvoirs publics un appui scientifique et technique notamment en situation d’alerte sanitaire, dans le cadre d’investigations de cas groupés humains («clusters») ou d’épidémies. Ils participent à la recherche des sources alimentaires ou environnementales responsables des contaminations humaines. Ils effectuent la comparaison de résultats d’analyse obtenus sur des isolats bactériens provenant de matrices issues de la chaîne alimentaire, de l’environnement ou au niveau des productions primaires.
Ces demandes d’analyses émanent de différents acteurs :
➤ Au niveau national :
• Santé publique France (SpF) : agence placée sous la tutelle du ministère de la santé en charge de la surveillance épidémiologique de la population humaine travaille en collaboration directe avec les CNR.
• La mission des urgences sanitaires (MUS) de la Direction générale de l’alimentation (DGAL)
➤ Au niveau Européen : l’ECDC (European Center for Disease Prevention and Control), en collaboration avec l’EFSA (European Food Safety Autority) assurent une surveillance épidémiologique harmonisée respectivement chez l’homme et sur la chaine alimentaire .
L’Anses résulte de la fusion, en juillet 2010, de l’agence française de sécurité sanitaire des aliments (Afssa) et de l’Agence française de sécurité sanitaire de l’environnement et du travail (Afsset). Placée sous la tutelle des Ministères chargés de la Santé, de l’Agriculture, de l’Environnement, du Travail et de la Consommation, l’agence a pour but de contribuer à assurer la sécurité sanitaire humaine, végétale et animale, à travers des domaines multiples. L’Anses compte neuf laboratoires en France, dont quatre sont spécialisés dans la sécurité des aliments et des eaux de consommation. Le laboratoire de sécurité des aliments (LSAl) intervient sur les dangers biologiques et chimiques qui peuvent affecter la sécurité sanitaire et la qualité des aliments. Il participe dans son domaine de compétence à l’accomplissement des missions de référence, de recherche, de veille, de surveillance et d’expertise scientifique et technique de l’Anses. Le LSAl se décline en plusieurs unités spécialisées dans les contaminants chimiques des aliments, dont une partie traite plus spécifiquement des produits de la pêche et de l’aquaculture, et les contaminants microbiologiques des aliments. Parmi ces unités, se situe l’unité Salmonella et Listeria (SEL). L’unité intervient donc dans l’analyse et le typage des deux pathogènes alimentaires, collectés à toutes les étapes « de la fourche à la fourchette ». Celle-ci exerce des activités dans le cadre de plusieurs mandats :
➤ LRUE Listeria monocytogenes
➤ LNR Listeria monocytogenes
➤ Laboratoire associé au LNR Salmonella (Anses de Ploufragan) .
L’unité SEL anime depuis 1997 un réseau de surveillance des salmonelles d’origine non humaine, le Réseau Salmonella (RS), constitué d’environ 150 laboratoires français volontaires, privés et publics, permettant de constituer une souchothèque et une base de données référençant les caractéristiques biologiques données épidémiologiques des isolats détectés dans les secteurs de la santé animale, l’hygiène des aliments et l’écosystème naturel. Ce réseau et apporte un appui technique en termes de typage des souches auprès des laboratoires partenaires. L’unité collecte des souches de L. monocytogenes dans le cadre des activités de LNR et LRUE pour ce pathogène. L’animation d’un réseau national similaire au RS sur Listeria monocytogenes est en cours d’étude. Ainsi, l’unité collecte, caractérise et type près de 20 000 souches par an sur les deux pathogènes confondus.
Les évènements sanitaires nécessitant une étude approfondie par typage peuvent être définis en trois grands groupes : les alertes produits, les alertes sanitaires et les toxi-infections alimentaires collectives (TIAC). En France, la coordination de ces déclarations se fait entre la MuS (gérée par le DGAL), SpF et les directions départementales en charge de la protection des populations(DDPP). Selon SpF, en France, les maladies infectieuses d’origine alimentaire représentent environ deux millions de cas annuels, environ 20 000 hospitalisations et 300 décès (Van Cauteren, 2017). Salmonella fait partie des principaux agents responsables des cas et des hospitalisations ; les infections à Salmonella spp. et Listeria monocytogenes représentent la moitié des décès d’origine alimentaire.
En cas d’apparition d’au moins deux cas similaires d’une symptomatologie pouvant être rapportée à une même origine, on parle de Toxi-Infections Alimentaires Collectives (TIAC). Elles constituent des maladies à déclaration obligatoire auprès des DDPP ou de l’Agence Régionale de Santé (ARS). Les TIAC sont souvent liées à l’utilisation de matières premières contaminées et/ou au nonrespect des mesures d’hygiènes et de température (rupture de chaîne du froid ou du chaud) lors des préparations culinaires. Le plus souvent, les TIAC sont familiales ou associées aux modes de restauration commerciale ou collective. Leur signalement entraine la réalisation d’une enquête épidémiologique destinée à identifier les aliments responsables, les facteurs favorisant leur contamination et de prendre des mesures correctives locales et rapides afin d’éviter la survenue de nouveaux cas de contamination.
|
Table des matières
1 Introduction
1.1 Les laboratoires français et européens impliqués dans la surveillance de Salmonella et Listeria monocytogenes
1.2 Le laboratoire de sécurité des aliments
1.3 Les différents types d’évènements sanitaires
1.4 Les microorganismes étudiés
1. Salmonella
2. Listeria monocytogenes
1.5 Le virage vers la génomique
1.6 La génomique en application
1.7 Assurance qualité
2 Présentation du projet
3 Sélection des panels
3.1 Salmonella enterica enterica Agona
3.2 Listeria monocytogenes CC204
4 Matériel et méthodes
4.1 Les étapes dites de « wet-lab »
4.2 Analyses génomiques, dites de « dry-lab
4.3 Détection des évènements de recombinaison
4.4 Comparaison des méthodes
4.5 Recherche des gènes de virulence, de résistance et de persistance
5 Résultats
5.1 Séquençage
5.2 Lancement des analyses bio-informatiques
5.2.1 S. Agona
5.2.2 L. monocytogenes CC204
5.3 Détection d’évènements de recombinaison
5.3.1 Le panel de S. Agona
5.3.2 Le panel de L. monocytogenes CC204
5.4 Comparaison des outils
5.4.1 Normalisation et contrôle qualité
5.4.2 Comparaison des matrices de distance par analyse de distance SNP et allélique
5.4.3 Comparaison des matrices de distances par le test statistique de Mantel
5.4.4 Comparaison des arbres phylogénétiques
5.4.5 Utilisation du script « matrix2association » permettant le clustering de souches dans le cadre d’alertes sanitaires
5.4.6 Comparaison des outils sur le plan paramétrique et fonctionnel
5.4.7 Recherche des gènes virulence, résistance et persistance
5.5 Valorisation des acquis
6 Conclusion
![]() Télécharger le rapport complet
Télécharger le rapport complet