Télécharger le fichier pdf d’un mémoire de fin d’études
PRINCIPALES RACES DE BOVINS EXPLOITEES AU SENEGAL
Il existe trois races bovines dominantes :
Le zébu Gobra (Bos indicus) encore appelé zébu peulh sénégalais. Il vit à l’ouest du Sénégal (Baol, Cayor, Djoloff), au nord du Sine-Saloum et le long du fleuve Sénégal, au sud de la Mauritanie et au nord ouest du Mali (MCD, 1991). Le Gobra est un bovin à bosse de grande taille (1,25 m à 1,46 m) et de poids à l’âge adulte variant de 350 kg à 450 kg chez le mâle et de 250 kg à 350 kg chez la femelle (SECK, 1993). Les cornes sont en forme de lyre haute, le fanon est très développé avec parfois des plis, sa tête est large, son front bombé, le chanfrein rectiligne, le chignon saillant, les oreilles longues et dressées, la robe est généralement blanche ou légèrement froment surtout chez le mâle où elle peut présenter des bringures et des charbonnures.
L’aptitude principale de cette race est la production de viande avec un poids de carcasse égal à 128,7 kg pour un rendement d’abattage de 56,7% chez les animaux âgés de 3 à 5 ans. Le poids de la carcasse atteint 373 kg avec un rendement d’abattage de 66,5 % chez les animaux extériorisés d’âge (BANKOLE et al., 1997).
Le taurin Ndama (Bos taurus) : on s’accorde à reconnaître au Ndama une très ancienne présence en Afrique vers les années 1534. Dans son berceau de race, le Fouta Djallon en Guinée, les bovins Ndama et leurs propriétaires peuls sédentaires sont les anciens descendants des occupants du Sahara (AKOUANGO et al., 2010). Il se caractérise par une absence de bosse au niveau du garrot, sa petite taille, des cornes en lyre, une robe qui présente toutes les nuances du fauve mais la couleur la plus répandue est le froment. Au Sénégal, le taurin Ndama est retrouvé au sud dans les régions de Tambacounda et de Casamance. C’est une race trypanotolérante (aptitude à vivre normalement dans un milieu naturel infesté, en hébergeant les trypanosomes pathogènes sans présenter de signes cliniques de la maladie.
Le Ndama est aussi une race à viande avec un poids à 4 ans qui peut atteindre 328,6 +-20,0 kg chez le mâle et 286,7+-8,3 kg chez la femelle avec un rendement carcasse de 50% en moyenne (SOW, 1987).
Le Djakoré est un métis naturel entre le zébu et le taurin. Il s’est développé dans la zone de contact entre les races Gobra et Ndama (nord du Sine-Saloum). Il s’agit d’une race dont les caractéristiques se rapprochent plus ou moins des races d’origine en fonction de sa situation géographique. Il présente une taille nettement supérieure à celle du taurin Ndama, une bosse peu marquée, le rein et le dos plats et larges, la ligne dorsolombaire rectiligne, le train postérieur musclé, la culotte bien descendue, les membres courts et puissants (BOYE et al., 2005).
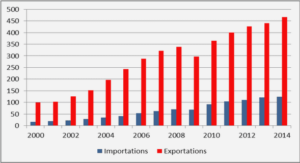
Ces bovins sont à la base de la production locale de lait qui est fortement tributaire des conditions climatiques ; en effet l’hivernage (juillet/octobre) est le moment propice pour une assez bonne production laitière mais cette production est très faible par rapport à celle des races exotiques car elle n’est que de 2 litres par vache locale (THIAM, 2005). De ce fait, cette production locale ne couvre pas la demande nationale d’où la nécessité des importations pour essayer de combler le déficit de l’insuffisance de l’offre locale (THIAM, 2005).
Pour combler ce déficit de production laitière et diminuer les importations, un programme d’insémination artificielle (IA), financé par la BAD (Banque Africaine de Développement) et le gouvernement du Sénégal, a été mis en œuvre en 1994 par le PAPEL (Projet d’Appui à l’Elevage) dans le bassin arachidier. Ce programme d’IA s’inscrit dans la dynamique de modernisation et d’intensification de systèmes de productions animales et sa zone d’emprise couvre presque tout le Sénégal. Et depuis 1994, ce programme d’IA suit son cours et après des tests concluants dans les années 1999-2000, des campagnes d’IA bovine se sont succédées de façon ininterrompue, depuis 2003, comme une activité phare dans le calendrier agricole.
SYSTEMES D’ELEVAGE
L’élevage s’intègre au niveau de trois systèmes (figure 2) à savoir :
le système pastoral : il concerne 32% des bovins et 35% des petits ruminants et est pratiqué au niveau de la zone sylvo-pastorale appelée le Ferlo situé dans la partie septentrionale du Sénégal. Cette zone constitue la plus grande zone d’élevage du Sénégal avec son relief, son climat, ses ressources hydrauliques végétales et animales très adaptés. Le Ferlo appartient aux domaines sahélien et soudano- sahélien avec un mode d’élevage extensif transhumant suivant les disponibilités fourragères (pâturages naturels) et hydriques (existence de points d’eau temporaires ou permanents et d’un réseau de forages profonds) (MBAYE et al 2006).
le système agro-pastoral : il concerne 67% et 62% des bovins et petits ruminants respectivement, et se caractérise par l’association élevage / agriculture (cultures pluviales avec le mil, l’arachide, le coton et cultures irriguées avec le riz, les tomates, les oignons). On le retrouve surtout dans le bassin arachidier, la vallée du fleuve Sénégal et la zone sud du pays. Cette association agriculture/ élevage se traduit généralement par la pratique de la culture attelée, l’utilisation de la fumure animale et l’exploitation des résidus de récolte pour alimenter le cheptel. Les modes de conduites des troupeaux y sont déterminées par la recherche de parcours saisonniers dans les limites des terroirs villageois ou à l’extérieur de la zone.
le système périurbain intensif : il concerne 1% de bovins et 3% des petits ruminants. Les modes d’élevage intensifs et semi intensifs y sont effectués et sont principalement présents dans la zone des Niayes. Cette zone des Niayes est aussi le siège d’un important élevage avicole qui intéresse de plus en plus les producteurs car très rentable.
MATERIEL ET METHODES
ECHANTILLONNAGE
Les trois races bovines locales à savoir le Gobra, le Ndama et le Djakoré au nombre de vingt-deux
(22) constituent les sujets de notre étude. Parmi ces races, treize (13) sont considérées comme pures
(F0), selon les éleveurs, réparties en 3 Djakoré, 7 Gobra et 3 Ndama et neuf (9) sont des métisses de première génération (F1) divisées en 5 Djakoré et 4 Gobra. Elles sont issues de deux zones agro écologiques du Sénégal à savoir le bassin arachidier (régions de Kaolack, Fatick et Kaffrine) et la zone sylvopastorale (région de Louga) et suivent toutes les systèmes d’élevage intensif ou semi-intensif. Les différentes localités de ces régions d’où proviennent ces animaux sont:
Kahone (14°41 de latitude nord et 16°25 de longitude ouest) dans la région de Kaolack (entre les longitudes 14°30 et 16°30 ouest et les latitudes 13°30 et14°30 nord)
Birkelane (14°09 de latitude nord et 15°45 de longitude ouest) dans la région de Kaffrine (13°50 de latitude nord et 15°25 de longitude ouest),
Mbadakhoune (14°09 de latitude nord et 14°11 de longitude ouest) et Santamba (13°43 de latitude nord et 16°25 de longitude ouest) dans la région de Fatick (14°19 de latitude nord et 16°25 de longitude ouest),
Dahra au niveau du CRZ (Centre de Recherches Zootechniques) dans la région de Louga (15°37 de latitude nord et 16°13 de longitude ouest).
Les informations, concernant les individus échantillonnés, à savoir leur identification, leur localité, leur catégorie, la couleur de leur robe, leur mode d’alimentation et leur utilisation sont consignés dans le tableau 3. L’identification des individus de bovins est faite en utilisant les deux premières lettres (la première lettre en majuscule et la seconde en minuscule) de la race suivies de la première lettre de la localité et de la lettre P si l’individu est de race pure et enfin, un chiffre qui détermine le numéro d’identification de l’animal. EXEMPLE : GoBP05 : 5ième individu de Gobra race pure de Birkelane et GoB03 :3ième individu de Gobra race métisse de Birkelane
Le tableau ci-dessous résume les données sur les animaux à partir desquels les 22 séquences d’ADN ont été obtenues avec leurs localités et leur statut supposé de pureté génique ou non avec F0 qui désigne les races pures et F1, les métisses de première génération.
ETUDE GENETIQUE
Afin de procéder à l’étude génétique à partir de l’ADN mitochondrial (ADNmt), le sang des bovins échantillonnés est prélevé au niveau de leur veine jugulaire et collecté dans des tubes avec EDTA (Acide Ethylène Diamine-Tétra-Acétique), l’EDTA permettant une meilleure conservation des acides nucléiques du sang. A la suite du prélèvement sanguin, l’extraction de l’ADNmt sera effectuée suivie de son amplification par PCR, de son séquençage et du traitement des données du séquençage grâce aux logiciels. Cependant, faisons un petit rappel sur l’ADN mitochondrial avec son gène, le cytochrome b.
L’ADN MITOCHONDRIAL (ADNmt)
L’ADNmt, avec son marqueur moléculaire le cytochrome b, est largement utilisé lors des analyses de la diversité génétique et phylogénétiques. L’ADNmt haploïde, transporté par les mitochondries du cytoplasme cellulaire, possède un mode de transmission maternelle d’hérédité, une absence de recombinaison génétique en général et un taux de mutation élevé qui permet de reconstruire les relations évolutionnaires intra et inter raciales. La variation de l’ADNmt est lente et se prête donc très bien au calcul de distance génétique sur des périodes relativement brève. Son principal marqueur, le cytochrome b, permet la détection rapide de l’hybridation entre les espèces et les sous espèces d’animaux d’élevage (NDIAYE, 2011).
EXTRACTION D’ADN
La méthode du Kit Qiagen DNeasy a été utilisée pour l’extraction de l’ADN à partir du prélèvement sanguin. Le protocole consiste à mélanger 100µl de sang anticoagulé à 20µl de protéinase K, afin de dégrader les protéines, auquel on ajoute du PBS (tampon phosphate salin) jusqu’à un volume de 220µl et on incube le tout à 70°C après y avoir mis 200µl de tampon AL. A la suite de l’incubation, 200µl d’éthanol sont versés dans le mélange et le tout est déposé dans une colonne DNeasy puis on procède à deux centrifugations du fait de la viscosité du sang pendant 1mn chacune à 13000 rpm (rotation par minute) pour retenir l’ADN dans la membrane de la colonne. En effet, l’ADN chargé négativement se fixe, par interactions ioniques, sur la membrane de silice chargée positivement alors que, les protéines, les lipides et les polysaccharides sont éliminés. L’effluent obtenu après centrifugation est jeté de même que le tube collecteur et la colonne est récupérée et mise dans un nouveau tube collecteur de 2ml dans lequel on dépose 500µl de tampon AW1. L’ensemble est centrifugé deux fois à 13000 rpm pendant 1mn et placé dans un nouveau tube collecteur avant d’y ajouter 500µl de tampon AW2 puis, de nouveau, une centrifugation est effectuée à 13000 rpm pendant 3mn. Ces deux tampons ont pour but de purifier l’ADN fixé et d’éliminer totalement toutes sources de contaminations. Afin de sécher la membrane DNeasy, on centrifuge encore pendant 2mn à 13000 rpm et on place la colonne dans un tube propre de 1,5ml pour éluer l’ADN dans 200µl de tampon AE préalablement chauffé. Pour finir, on procède à une incubation pendant 1mn suivie d’une centrifugation à 13000 rpm pendant 1mn et d’une conservation de l’ADN élué à -20°C.
L’AMPLIFICATION PAR PCR (réaction de polymérisation en chaîne)
La PCR est une technique de la biologie moléculaire qui consiste à amplifier une portion spécifique d’un gène dont on connait la séquence. Cette amplification a été rendue possible grâce à la découverte d’une ADN polymérase (la Taq polymérase) capable de résister aux températures de dénaturation. Cette enzyme est capable à partir d’amorces spécifiques d’ajouter des nucléotides de façon complémentaire et antiparallèle au gène dont on désire l’amplification (GUERIN et al., 1993).
L’amplification de l’ADNmt est effectuée dans un thermocycleur suivant le protocole classique de PCR avec deux amorces du gène de cytochrome b à savoir le H15915 (5’-TCT CCA TTT CTG GTT TAC AAG AC-3’) et le L14723 (5’-ACC AAT GAC ATG AAA AAT CAT CGT T-3’). Ces amorces sont spécifiques aux rongeurs et amplifient des séquences de 1140 paires de bases.
Le volume réactionnel pour la PCR est de 25µl constitués de 10.8µl d’eau MilliQ, de 2.5µl de tampon PCR (à 10fois), de 1.5µl de MgCl2 supplémentaire à 25mM, de 1µl dNTPs à 25mM, de 2.5µl de H15915 à 10µM, de 2.5µl de L14723 à 10µM, de 0.2µl de Taq polymérase à 5U/µl et 4µl d’ADN dilué au 1/10.
A la suite de ce mélange, l’amplification est réalisée suivant les différentes étapes du protocole PCR pour l’obtention de 40 cycles au final. Ces étapes se résument à une dénaturation initiale à 94° C pendant 3mn suivie d’une seconde dénaturation toujours à 94°C pendant 45 secondes puis surviennent les étapes d’hybridation à 48°C pendant 1mn et d’élongation à 72°C pendant 1mn 30 secondes. Une élongation finale à 72°C pendant 10mn met fin au processus et on ramène la température à 10°C pour permettre un refroidissement du produit avant de procéder au séquençage du gène.
SEQUENCAGE DU GENE DE CYTOCHROME B
Ce séquençage, qui s’est effectué en Corée du Sud dans la ville de Séoul par la société MACROGEN, permet de déterminer l’ordre d’enchainement des séquences de nucléotides d’un fragment d’ADN. C’est une réaction de PCR particulière utilisant, en plus des composés habituels (ADN matrice, polymérase, amorces, dNTP, Mg2+), des nucléotides modifiés les didésoxyribonucléotides ou ddNTP.
ANALYSES GENETIQUES DES DONNEES DU SEQUENCAGE
Trois logiciels sont utilisés dans le cadre de cette étude à savoir, le logiciel BioEdit (version 7.0.5.3) permettant aussi bien d’ouvrir, de nettoyer et d’aligner des séquences ainsi que de transformer des séquences nucléotidiques en séquences protéiques suivi du logiciel DNAsp (version 5.10.01) avec lequel nous pouvons déterminer les sites conservés ou variables, les singletons, les sites de parcimonie informative de l’ensemble des jeux de données, le nombre d’haplotypes, la diversité haplotypique et nucléotidique de l’ensemble des séquences et la divergence génétique des populations et enfin, le logiciel MEGA5 (version 5) qui permet non seulement la détermination des pourcentages de transitions et de transversions et des distances génétiques intra et inter populationnelles mais aussi la reconstruction des arbres phylogénétiques avec les trois approches possibles (Neighbor-Joining, Maximum de Parcimonie et Maximum de Vraisemblance). Et pour ces trois reconstructions, le test de bootstrap a été utilisé afin de témoigner de la robustesse des nœuds internes qui représentent les ancêtres communs des taxons ; de ce fait, une valeur de booststrap au-delà de 50% montre la solidité des liens de parenté entre les différents individus concernés.
|
Table des matières
INTRODUCTION
CHAPITRE I : ETUDE BIBLIOGRAPHIQUE
I. PRESENTATION DU SENEGAL
I.1. SITUATION
I.2. POPULATION
I.3. RELIEF
I.4. CLIMAT
II. ELEVAGE AU SENEGAL
II.1. CHEPTEL
II.2. LES NOTIONS DE RACE – RACE PURE EN ELEVAGE ET EN GENETIQUE
II.3 PRINCIPALES RACES DE BOVINS EXPLOITEES AU SENEGAL
II.4. SYSTEMES D’ELEVAGE
CHAPITRE II: MATERIEL ET METHODES
I. ECHANTILLONNAGE
II. ETUDE GENETIQUE
II.1. L’ADN mitochondrial (ADNmt)
II.2. Extraction d’ADN
II.3. L’amplification par PCR (réaction de polymérisation en chaîne)
II.4. Séquençage du gène de cytochrome b
II.5. Analyses génétiques des données du séquençage
CHAPITRE III : RESULTATS ET DISCUSSION
I.RESULTATS
I.1. LES SEQUENCES ALIGNEES
I.2. La diversité génétique des populations de races bovines
I.2.1. La variabilité génétique
I.2.2. La diversité génétique entre les populations de bovins
I.2.3. les distances génétiques intra et inter raciales des populations de bovins
I.2.4.Les pourcentages de transitions et de transversions
I.3. LES RECONSTRUCTIONS PHYLOGENETIQUES
II. DISCUSSION
CONCLUSION
BIBLIOGRAPHIE
Télécharger le rapport complet