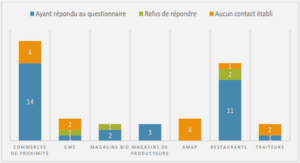
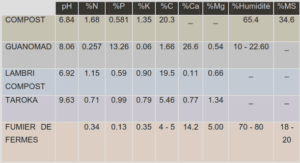
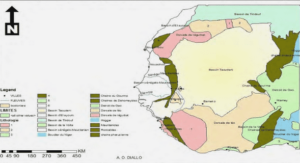
Télécharger le fichier pdf d’un mémoire de fin d’études
Les clés d’identification à accès simple
Fonctionnement et particularités des clés d’identification à accès simple
Une clé d’identification à accès simple peut prendre plusieurs formes, mais elle est toujours constituée d’une série d’étapes que l’utilisateur doit suivre pour aboutir à une identification de son spécimen. On la dit ‘’à accès simple’’ car cette suite d’étapes est fixée pour une même clé d’identification. L’ordre des étapes successives est imposé (Figure 7). À chaque étape de la clé, plusieurs alternatives sont proposées à l’utilisateur, qui doit sélectionner celle qui correspond au spécimen qu’il souhaite identifier. Le choix d’une alternative détermine l’étape suivante, et ainsi de suite, jusqu’à aboutir à une identification finale. Si deux alternatives exactement sont proposées à chaque étape – par exemple, ‘’présence d’ailes’’ versus ‘’absence d’ailes’’, ou encore ‘’pattes de couleur rouge’’ versus ‘’pattes de couleur jaune’’ – la clé est dite dichotomique. Si trois alternatives ou plus sont proposées à chaque étape – par exemple, feuille ‘’lancéolée’’ ou ‘’ovale’’ ou ‘’palmée’’ ou ‘’pennée’’ ou ‘’réniforme’’ – la clé est alors dite polytomique.
Les clés d’identification à accès simple telles qu’elles sont représentées dans les ouvrages ne suivent pas toujours la systématique des taxons d’intérêt ni leur classification phylogénétique au sein d’un groupe plus large d’être vivants. Elles ne renseignent pas forcément sur les relations évolutives entre organismes. La discrimination progressive entre les différents objets inclus dans une clé donnée se fait à chaque étape du chemin d’identification, selon des critères discriminants arbitraires propres aux objets décrits et à cette clé d’identification en particulier. L’auteur de la clé fait le choix des critères distinctifs de sorte à réduire le nombre d’étapes nécessaires à l’obtention d’une identification pour l’objet étudié, et privilégie les caractères les moins ambigus possibles à chaque étape du chemin.
A l’origine des clés d’identification à accès simple
Depuis le début de leur existence, les clés de détermination peuvent traiter de toutes sortes d’éléments – y compris en-dehors des sciences naturelles – à partir du moment où ces derniers sont reconnaissables et caractérisables grâce à différents critères. Parmi les premières clés d’identification connues, celles de Petrus Ramus (1515–1572), logicien et philosophe français, étaient dichotomiques et portaient sur la dialectique (Ramus 1574) (Figure 8a) ou encore sur les opérations arithmétiques (Ramus 1592, Ong 1958). La volonté de Ramus était de mettre à disposition des informations de façon claire et compréhensible, afin qu’elles puissent être enseignées et soumises au débat (Griffing 2011). Dès leurs origines, les clés dichotomiques se voulaient spécifiquement une aide à l’apprentissage, basée sur la découverte et sur une simplification des idées pour les mettre à portée du plus grand nombre.
John Ray (1627–1705), naturaliste anglais membre de la Royal Society, publie en 1686 la première partie de son Historia plantarum generalis (Ray 1686), qui peut être considérée comme la première flore mondiale. L’ouvrage possède des clés d’identification dichotomiques (Figure 8b). Cependant, l’absence de schémas ou de dessins au sein de ces clés met rapidement en lumière l’importance des illustrations pour l’étude des objets de sciences naturelles. Ray inclut donc ensuite de nombreux dessins à l’aquarelle dans son ouvrage intitulé Synopsis methodica stirpium Britannicarum (Ray 1690). De même Richard Waller (1650–1725), un autre naturaliste anglais membre de la Royal Society, publie ses Tables of the English herbs (Waller 1689), une clé d’identification dichotomique richement illustrée au crayon et à l’aquarelle (Figure 9). Comme Ramus, la volonté de Waller est de s’adresser à tous (y compris aux ‘’novices’’) grâce à la simplification de certaines classifications botaniques, et surtout à ses nombreuses illustrations, au lieu de se reposer uniquement sur du texte pour ses descriptions (Griffing 2011).
Types de caractères utilisés dans les clés
La grande majorité des clés d’identification repose sur l’utilisation de caractères morphologiques ou anatomiques pour décrire les spécimens. Ces types de descripteurs sont relativement simples à coder en tant que descripteurs discrets, par exemple ‘’présence’’ vs ‘’absence’’ ou ‘’pattes noires’’ vs ‘’pattes jaunes’’ vs ‘’pattes orange’’.
Cependant, d’autres types de critères peuvent être utilisés, seuls ou en complément des structures morpho-anatomiques. Si l’utilisateur de la clé a accès à ces informations, il est possible d’inclure dans les clés des traits relatifs à l’écologie ou à l’histoire de vie des organismes. Il peut s’agir par exemple de caractéristiques de leur milieu de vie (localisation géographique, pH et température de l’eau pour les organismes aquatiques, habitat ombragé ou non…) ou de particularités biologiques et comportementales (période de ponte ou de floraison, mode de déplacement, régime alimentaire…). La clé réalisée par Zervakis et al. (2014) a l’originalité de prendre en compte les associations entre des champignons et des plantes. Certaines clés numériques réalisées sous Xper3 (la plateforme que j’ai utilisée pour réaliser des clés d’identification) intègrent des critères écologiques. Parmi elles, nous pouvons noter la clé ‘’Insectes’’ du Spipoll44 (présentée en détails au 4.2.2a), une clé des orchidées de France45 (Dubuisson et al. 2018), une clé des guêpes du genre Pteromalus (Klimmek & Baur 2018) et une clé des chouettes et hiboux de France46.
Certaines clés d’identification sont basées sur des critères sonores, par exemple les chants, cris ou sons émis par des animaux. Dans certains cas, il est possible de coder des caractères simples tels que la durée, la hauteur, la fréquence ou les patrons de répétition d’un son. Cela a déjà été réalisé, par exemple pour certains oiseaux, chauve-souris et criquets (Reinhold et al. 2001, Warren 2003, Szövényi et al. 2012, Iorgu et al. 2017). Les clés de détermination en ligne permettent souvent aussi d’intégrer des fichiers audio, qui peuvent servir à la description et/ou à vérifier la justesse de l’identification. C’est le cas de la clé ‘’Key to garden and village birds’’47 qui combine une description écrite des sons produits par les oiseaux et des fichiers audio permettant d’écouter un court extrait des chants, cris et sons des oiseaux (Trilar 2010).
Des clés d’identification moléculaires basées sur des séquences d’ADN (produits de PCR notamment) existent également, pour des organismes aussi divers que l’olivier, des levures, des insectes thysanoptères, des annélides polychètes et des moules (Gerke & Tiedemann 2001, Cadez et al. 2002, Rugman-Jones et al. 2006, Blank et al. 2008, Alba et al. 2009).
Clés numériques à accès simple
Depuis la généralisation de l’informatique, les clés à accès simple se retrouvent également ailleurs que sur des supports papier. Ce type de clés peut ainsi être numérisé, et accompagné si besoin de supports tels que des illustrations, des fichiers audio et vidéo, des liens URL renvoyant vers des pages web d’intérêt. C’est le cas de la clé d’identification des coccinelles disponibles sur le site web ‘’Discover Life’’48, et de la clé générée à partir de la base de données Perla pour les ‘’invertébrés d’eau douce’’49. Pour faire un choix à chaque étape du chemin d’identification, l’utilisateur doit cliquer sur le lien web correspondant à la proposition qu’il estime juste (Figure 10), ce qui l’amène à l’étape suivante de la discrimination.
Les clés d’identification numériques à accès libre
Principes et caractéristiques
Depuis les années 1960, l’informatique a grandement aidé les biologistes dans leurs travaux pour la construction de clés d’identification. Des méthodes d’identification assistée par ordinateur (IAO) ont vu le jour pour permettre la réalisation de clés plus souples, dites interactives, et pour les diffuser sur le web. Parmi ces IAO, nous pouvons citer les logiciels ‘’Online’’ (Pankhurst & Aitchinson 1975), Xper (Lebbe 1984), DELTA et IntKey (Dallwitz 1993).
Les clés d’identification interactives à accès libre sont des outils numériques d’aide à l’identification. Elles ne suivent pas un cheminement imposé comme c’est le cas pour les clés à accès simple (arbre de décision). L’utilisateur choisit lui-même les critères qu’il va utiliser pour la description de son spécimen, et l’ordre dans lequel il les utilise (Figure 11). Il n’y a pas de limite au nombre de taxons, de caractères et d’états de caractères qui peuvent être inclus dans la clé de détermination. Les caractères numériques peuvent être codés directement, sans être divisés en catégories (l’utilisateur peut indiquer un nombre) (Dallwitz et al. 2013). Les données sont codées sous la forme d’une matrice taxons-caractères. Il est possible de mettre en place des dépendances, c’est-à-dire des caractères qui ne sont applicables que si certaines valeurs précises ont été sélectionnées pour un autre caractère donné – ils sont appelés ‘’descripteurs-fils’’ dans Xper3.
L’utilisateur peut également revenir sur ses choix pour les modifier, ainsi que sélectionner plusieurs réponses possibles s’il doute à une étape. Il est possible de configurer l’algorithme de telle sorte qu’une identification correcte peut être réalisée malgré une ou plusieurs ‘’erreurs’’ de la part de l’utilisateur au cours de la détermination. De manière générale, l’un des objectifs principaux des clés à accès libre est de réduire le risque d’obtenir une mauvaise détermination à la fin de la démarche d’identification.
Pourquoi ce type de clés ? Avantages et inconvénients des clés d’identification numériques à accès libre
Les clés à accès simple présentent un certain nombre d’inconvénients et limites. Certains d’entre eux peuvent être contournés ou réduits par l’utilisation d’outils d’identification numériques à accès libre. Le Tableau 1 résume les principales limites des clés à accès simple et les caractéristiques des clés à accès libres qui permettent de pallier ces inconvénients.
De manière générale, les clés d’identification informatisées interactives apportent une facilité d’utilisation et une flexibilité supérieures à celles des clés à accès unique (Edwards & Morse 1995). Cependant, elles apportent leur lot de difficultés :
L’abondance de critères de détermination proposés et la liberté de choix de l’utilisateur rend le nombre d’étapes d’identification, et donc le temps passé pour effectuer la détermination, très variables selon l’utilisateur et l’état du spécimen à décrire – et souvent supérieurs à l’optimal
L’absence de chemin d’identification imposé et l’abondance de possibilités peut rendre plus difficile sa tâche à l’utilisateur, qui peut se sentir perdu ou submergé
La possibilité de sélectionner plusieurs choix aux étapes d’identification fait que plusieurs propositions finales de taxons peuvent être conservées ; l’utilisateur n’obtient pas alors d’identification unique.
L’apport des clés numériques à accès multiples pour les sciences participatives : l’exemple du Spipoll
Le programme Spipoll (Suivi Photographique des Insectes POLLinisateurs) est un projet de sciences participatives ayant pour but de collecter sur l’ensemble de la France métropolitaine et selon un protocole défini, des observations d’insectes pollinisateurs et/ou floricoles. Ces données sont ensuite utilisées à des fins scientifiques d’analyse des variations de diversité, de densité et de structure des réseaux de pollinisation sur le territoire. Depuis sa création en 2010, des articles scientifiques utilisant les données récoltées par le programme Spipoll ont été publiés dans des revues internationales à comité de lecture (Deguines et al. 2012, Shwartz et al. 2013, Deguines et al. 2016, Desaegher et al. 2018, Levé et al. 2019).
Les volontaires effectuent des ‘’sessions d’observation’’ qui durent au moins 20 minutes (durée minimale requise dans le protocole) mais qui peuvent être prolongées si le volontaire le désire. Ce dernier choisit une espèce de plante à fleurs (plantée par l’homme ou non), prend en photo la plante et son environnement, note des paramètres environnementaux (température, milieux de vie proches, intensité et régularité du vent, ensoleillement…) ainsi que la date et l’heure de la session d’observation. Pendant l’intégralité de la session, le volontaire photographie tous les insectes qu’il voit se poser sur les fleurs de l’espèce qu’il a choisie. Ensuite, il trie ses photos pour garder un cliché par type d’insecte observé, et dépose ses données sur le site web du programme50. L’ensemble ‘’photos de la fleur + photo de l’environnement + une photo par type d’insecte qui s’est posé sur une fleur’’ est appelé ‘’collection’’ (Figure 12).
Taxons étudiés et logiciels utilisés
Échantillonnage taxonomique pour l’entomofaune
Les clés dites ‘’des larves’’ (T1, T2 et T4) traitent uniquement de la morphologie des stades larvaires les plus développés (également appelés ‘’nymphes’’), précédant la mue imaginale. Contrairement aux stades plus jeunes (pas complètement développés), la nymphe présente toutes les caractéristiques morphologiques observables au niveau larvaire, et son observation permet donc sans ambiguïté de discriminer chaque spécimen à l’espèce.
Les clés dites ‘’des imagos’’ (T3, T5, T6 et T7) concernent les individus ‘’adultes’’ ayant effectué leur dernière mue et possédant leurs couleurs et morphologie finales. Les ‘’subimagos’’ ne sont pas traités.
Insectes aquatiques de la région Ile-de-France
Pour les insectes aquatiques – odonates, éphémères, plécoptères et coléoptères, correspondant aux bases de données T1 à T6 – j’ai inclus toutes les espèces connues (ou supposées) comme étant présentes, au moins à un moment de leur cycle de vie, dans la région Ile-de-France. La liste d’espèces provient principalement de données récentes (depuis 1950) avec des occurrences vérifiées sur le terrain, fournies par les structures et programmes suivants : l’Office Pour les Insectes et leur Environnement (OPIE)52, la base de données du Cettia-idf (observatoire francilien de la biodiversité)53, et l’Inventaire national du patrimoine naturel (INPN)54.
Quelques espèces ont été ajoutées sur les conseils d’experts des taxons, qui assument qu’elles pourraient être rencontrées en Ile-de-France, sachant qu’elles sont présentes dans un ou plusieurs département(s) limitrophe(s) de la région (Michel Brulin, Jacques Le Doaré, comm. pers.).
Les ordres traités ont été choisis sur la base des ressources disponibles pour la création de bases de données, de l’intérêt pour la conservation des milieux aquatiques, et de la faisabilité sur un travail de 3 ans. Il s’agit d’ordres utilisés pour des inventaires de biodiversité dans le cadre de la Directive européenne Cadre sur l’Eau (voir 1.3.1). Le nombre de taxons reste raisonnable pour la faisabilité d’un travail exploratoire de thèse, et une quantité assez conséquente d’informations est disponible pour permettre la construction d’une clé d’identification juste et fiable basée sur la morphologie.
La priorité a été donnée aux larves d’odonates par rapport aux imagos, dû au manque d’outils et de guides d’identification pour les larves, qui sont beaucoup moins connues.
Seuls les coléoptères adultes (imagos) ont été inclus, car les connaissances sont très lacunaires pour les larves, et pour certaines espèces la correspondance taxonomique entre larve et adulte n’est pas connue. La clé d’identification des coléoptères aquatiques est la seule à proposer une détermination au genre et pas à l’espèce, compte tenu du nombre important d’espèces présentes en Ile-de-France (environ 250).
Les bases de données des insectes aquatiques réalisées pendant cette thèse sont donc ainsi composées :
Odonates : 59 espèces (22 zygoptères et 37 anisoptères) appartenant à 28 genres et 9 familles (Clé T1, Tableau 2)
Éphémères : 39 espèces appartenant à 21 genres et 9 familles (Clés T2 et T3, Tableau 3)
Plécoptères : 10 espèces appartenant à 8 genres et 5 familles (Clés T4 et T5, Tableau 4)
Coléoptères aquatiques : 68 genres appartenant à 15 familles (Clé T6, Tableau 5).
Coccinelles (imagos) de France métropolitaine
L’objectif des clés construites au cours de ce doctorat était qu’elles puissent être aisément appréhendées par tous les types de publics adultes, en utilisant des caractères visibles à l’œil nu ou à l’aide d’une loupe binoculaire. De ce fait, nous avons restreint l’échantillonnage taxonomique aux tribus Chilocorini, Coccinellini and Epilachnini. Les membres de ces tribus sont relativement grands (3 à 9 mm) et présentent une importante diversité de colorations, ce qui les rend facilement visibles dans leur environnement et identifiables par des non-spécialistes. Nous avons également inclus dans l’échantillonnage (sous forme d’items dans la clé d’identification) les formes de colorations les plus courantes, afin de couvrir au mieux les variabilités intraspécifiques de ces espèces.
La liste d’espèces est tirée de Tronquet (2014) et la taxonomie suit Seago et al. (2011). Pour plus de détails concernant les choix faits pour l’échantillonnage taxonomique, se référer à l’article correspondant (Jouveau & Delaunay et al. 2018 – Annexe 1). La plupart des spécimens ont été examinés dans les collections du MNHN.
La base de connaissances des coccinelles de France réalisée pendant cette thèse comprend donc 47 espèces appartenant à 24 genres et 3 tribus, tous inclus dans la sousfamille Coccinellinae et la famille Coccinellidae (Clé T7, Tableau 6).
|
Table des matières
Préambule : contexte de la thèse
Chapitre 1 – Introduction
1.1. Décrire, identifier et étudier la biodiversité
1.1.1. État et enjeux de la biodiversité
1.1.2. Comment décrire et identifier la biodiversité ?
1.1.3. Les sciences participatives : l’implication des citoyens pour l’étude de la biodiversité. 7
1.2. Le cas particulier de l’entomofaune
1.2.1. Les insectes : des organismes clés très diversifiés mais en fort déclin
1.2.2. Les difficultés rencontrées dans l’identification des insectes
1.3. L’entomofaune étudiée dans ce travail de thèse : des organismes d’intérêts variés
1.3.1. Les insectes aquatiques de la région Ile-de-France
1.3.2. Les coccinelles de France métropolitaine
1.3.3. Les insectes pollinisateurs de France métropolitaine
1.4. Objectifs et problématiques de la thèse
Chapitre 2 – Caractéristiques et historique des clés d’identification
2.1 Les clés d’identification à accès simple
2.1.1 Fonctionnement et particularités des clés d’identification à accès simple
2.1.2 A l’origine des clés d’identification à accès simple
2.1.3 Types de caractères utilisés dans les clés
2.1.4 Clés numériques à accès simple
2.2 Les clés d’identification numériques à accès libre
2.2.1 Principes et caractéristiques
2.2.2 Pourquoi ce type de clés ? Avantages et inconvénients des clés d’identification numériques à accès libre
2.2.3 L’apport des clés numériques à accès multiples pour les sciences participatives : l’exemple du Spipoll
Chapitre 3 – Comment construire des clés d’identification numériques ?
3.1 Taxons étudiés et logiciels utilisés
3.1.1 Échantillonnage taxonomique pour l’entomofaune
3.1.2 Xper, une suite d’outils pour la construction et l’analyse de bases de données et de clés d’identification
3.2 Méthodologie pour la construction de clés d’identification numériques sous Xper3
3.2.1 Modèle descriptif
3.2.2 Caractères morpho-anatomiques
3.3 Construction de clés Xper3 pour les insectes dans le cadre de la thèse
4.1 Évaluation du contenu des clés d’identification
4.1.1 Contenu général des clés d’identification
4.1.2 Pouvoir discriminant des descripteurs
4.2 Évaluation de l’utilisation des clés d’identification
4.2.1 Le système Xperience
4.2.2 Analyse des démarches d’identification pour la clé Spipoll
4.2.3 Test de l’utilisation de la clé des larves d’odonates d’Ile-de-France
Chapitre 5 – Comment améliorer des clés d’identification numériques ?
5.1 Intégration de critères écologiques dans les clés d’identification
5.1.1 Le cas des larves d’insectes aquatiques : odonates et éphémères
5.1.2 L’apport des critères écologiques
5.1.3 Une étude moléculaire pour affiner les critères écologiques
5.2 Prise en compte de l’expérience des utilisateurs des clés d’identification
5.2.1 Pour la clé ‘’Insectes’’ du Spipoll
5.2.2 Pour la clé des larves d’odonates d’Ile-de-France
5.3 Questionnaire et expérience des volontaires au Spipoll
5.3.1 Contexte
5.3.2 Matériel et méthodes
5.3.3 Résultats et discussion
5.3.4 Pistes d’amélioration de la clé d’identification du Spipoll
Chapitre 6 – Discussion générale et conclusion
6.1 Bilan du travail de thèse
6.2 De nouvelles méthodes d’identification
6.2.1 Les logiciels d’identification automatique
6.2.2 L’identification par séquençage rapide d’ADN
6.3 L’utilisation de critères non morphologiques dans les clés d’identification
6.4 Conclusion
RÉFÉRENCES
Télécharger le rapport complet