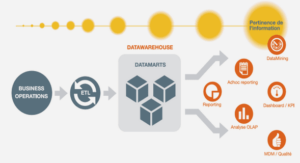
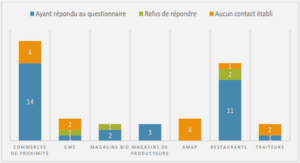
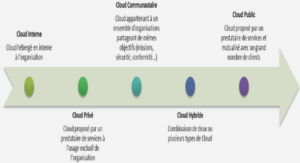
Télécharger le fichier pdf d’un mémoire de fin d’études
Bref historique des modèles de croissance de plantes
Très tôt, l importance capitale des plantes a poussé l homme à étudier leurs caractéristiques, d abord d un point de vue botanique, depuis Aristote et l un de ses disciples éophraste, à qui l on doit le plus ancien traité de botanique, puis agronomique, notamment grâce à Olivier de Serres et son ouvrage pionnier éâtre d’agriculture publié en 1600.
Les premiers modèles de croissance de plantes sont, eux, beaucoup plus récents, et remontent au début des années 1970. Depuis, ils n ont cessé de gagner en précision et complexité grâce aux progrès constants de l informatique. Nous présentons ici un bref aperçu de l histoire des modèles de croissance de plantes, avec d abord les modèles géométriques d un côté et agronomiques de l autre, puis l approche récente consistant à combiner ces deux types d approches dans ce que l on appelle les modèles structure-fonction.
D un point de vue botanique, la structure modulaire des plantes sous la forme d une succession de métamères a permis l émergence de modèles architecturaux, dont l objectif est de classer les végétaux en fonction de leurs modes de développement. Dans les années 1970, les botanistes Hallé et Oldeman pro-posent notamment un système de classiication permettant de répartir toutes les espèces d arbre connues en 23 catégories, en fonction de leur mode de croissance, de ramiication, de la différentiation morpholo-gique ou de la position des organes reproducteurs Hallé et Oldeman, 1970 ; Hallé et al., 1978 . Puis, dans les années 1980, l avènement de l informatique a permis le développement de modèles entièrement basés sur la simulation. Parmi eux, on retrouve les L-systèmes, introduits par Lindenmeyer à la in des années 1960, d abord pour décrire la croissance d organismes multicellulaires Lindenmayer, 1968 , puis appli-qués plus tard à la croissance des plantes dans Prusinkiewicz et al. 1988 voir aussi l ouvrage de référence e Algorithmic Beauty of Plants Prusinkiewicz et Lindenmayer, 1990 . Plusieurs extensions ont ensuite été proposées, notamment les L-systèmes stochastiques ou les grammaires relationnelles Kurth, 1994 , et de nombreux logiciels basés sur ces grammaires formelles ont vu le jour depuis les années 1990 : L-studio Federl et Prusinkiewicz, 1999 ; Karwowski et Prusinkiewicz, 2004 , GroIMP Kniemeyer et al., 2007 , . . . Une autre approche est celle développée au CIRAD ² dans les modèles AMAP de Reffye et al., 1988 ; de Reffye et Dinouard, 1990 ; de Reffye et al., 1991 . Cependant, si ces modèles permettent d obtenir une représentation idèle de l architecture de la plante, ils ne permettent pas de prendre en compte l interaction avec le fonctionnement Vos et al., 2007 ³.
Parallèlement au développement de ces modèles architecturaux, des modèles agronomiques ou « process-based » ont émergé, avec pour objectif de quantiier la production végétale au niveau du mètre carré, en fonction des conditions environnementales. Dans ce type de modèle, l architecture de la plante n est pas prise en compte, celle-ci étant simplement divisée en plusieurs compartiments d organes feuilles, tiges, racines, fruits, . . . . La production de biomasse s obtient ensuite grâce à un système d équations mettant en jeu les processus biologiques de photosynthèse, respiration, allocation, … En général, la quantité de rayonnement reçue par la plante y est modélisée par l intermédiaire de la loi de Beer-Lambert de Wit et al., 1970 ; Monteith, 1977 , empruntée à la physique optique, et qui permet de relier la quantité de lu-mière absorbée à l épaisseur du milieu traversé. Ces modèles peuvent être spéciiques à une espèce donnée par exemple, CERES-MAÏZE Jones et Kiniry, 1986 , ou génériques PILOTE, Mailhol et al., 1996 , et peuvent prendre en compte un grand nombre de processus écophysiologiques STICS, Brisson et al., 1998 . S ils permettent en général une bonne estimation du rendement des cultures, plusieurs auteurs Le Roux et al., 2001 ; Kurth, 1994 ont montré qu une prise en compte de l architecture permettrait d aug-menter leurs performances, à cause de l interaction forte qui existe entre la structure de la plante et son fonctionnement.
C est au carrefour de ces deux pratiques que sont nés les modèles de type structure-fonction, avec la première conférence internationale Funtional-Structural Plant Growth Models sur le sujet en 1996 Kor-pilaht, 1997 voir aussi Sievänen et al., 2000 pour une revue détaillée . Ces modèles permettent de combiner la description du développement de la structure de la plante au cours du temps, et les processus éco-physiologiques mis en jeu photosynthèse, respiration, allocation , eux-mêmes dépendants des condi-tions environnementales. Deux approches sont alors possibles pour construire ce type de modèles : soit étendre les modèles architecturaux en y ajoutant le fonctionnement de la plante, soit raffiner les modèles agronomiques ain de prendre en compte l architecture de la plante. La première approche a mené par exemple à la création du langage L+C Karwowski et Prusinkiewicz, 2003 , basé sur une extension des L-systèmes, ou encore à la création du modèle Greenlab, initié au LIAMA par de Reffye et Hu 2003 comme une suite logique des modèles AMAP. Basé sur un pas de temps discret le cycle de croissance , le modèle permet de déterminer à chaque cycle le nombre d organes créés, puis en déduit la production de biomasse par photosynthèse et l allocation de cette biomasse aux organes existants ou nouvellement créés. La deuxième approche a abouti par exemple à la construction du modèle LIGNUM Perttunen et al., 1996 , dont le fonctionnement est proche de celui du modèle Greenlab.
Greenlab
Allocation
– entre 0 et τ1, la graine n a pas encore commencé à germer, rien ne se passe
– entre τ1 et τ2, la plante produit ses premières feuilles et racines à partir de la graine
– à partir de τ2, la plante produit sa biomasse par photosynthèse.
Dans la suite, nous notons poal le vecteur de paramètres d allocation de l organe o, et pal = (poal)o∈O le vecteur contenant tous les paramètres d allocation de tous les organes.
Méthodes basées sur l’estimation des paramètres individuels
Méthodes basées sur une approximation de la vraisemblance
Lorsque le nombre dobservations par individu est trop faible, et comme cest le cas dans la plupart des problèmes non linéaires, une autre approche consiste à linéariser la fonction g, et à appliquer ensuite les méthodes disponibles dans le cas linéaire. Plusieurs méthodes ont été proposées, parmi lesquelles la méthode FO First-Order approximation introduite par Beal et Sheiner 1982, et la méthode FOCE First-Order Conditional Estimation introduite par Lindstrom et Bates 1990. Dans la méthode FO, Beal et Sheiner 1982 proposent un développement de Taylor dordre 1 autour du point ξi = 0, en supposant le terme ξiεij négligeable . La fonction g ainsi linéarisée, la densité conditionnelle f(y | ϕi; θ) peut donc être approchée par la densité dun vecteur gaussien dont lespérance et la matrice de covariance dé- pendent linéairement de ϕi. Lorsque la densité f(ϕi; θ) est également gaussienne, lintégrale 2.5 devient calculable analytiquement. Cette méthode est notamment implémentée dans le logiciel NONMEM®, principalement utilisé dans le domaine de la pharmacodynamique et de la pharmacocinétique, et dans la procédure NLMIXED de SAS®. Néanmoins, lapproximation FO peut savérer médiocre, et fournir des résultats biaisés Vonesh, 1992 ; Davidian et Giltinan, 1995.
Dans la méthode FOCE, proposée par Lindstrom et Bates 1990 comme une amélioration de la mé- thode FO, la linéarisation du modèle ne se fait plus autour du point ξi = 0, mais autour du mode a posteriori de ξi, correspondant au meilleur prédicteur linéaire non biaisé dans le cas du modèle linéaire mixte. Elle est également implémentée dans NONMEM® et dans SAS®, ainsi que dans la fonction nlme de R. Si cette méthode fournit de meilleurs estimateurs que la méthode FO Vonesh, 1992, lapproximation sur laquelle elle repose peut savérer mauvaise, en particulier lorsque lhypothèse de normalité nest pas vériiée.
Lalgorithme EM est un algorithme itératif dont lobjectif est dobtenir le maximum de vraisemblance dun modèle dans lequel certaines données ne sont pas observées et sont donc considérées comme manquantes. Il est particulièrement adapté aux cas où la vraisemblance des données complètes sécrit plus simplement que la vraisemblance des données observées, et repose sur lidée suivante : lorsque lon se trouve en présence de données manquantes, une première intuition est destimer ou de remplacer ces données manquantes, puis destimer les paramètres du modèle à laide des données « augmentées ». Chaque itération de lalgorithme se divise alors en deux étapes, lune dite « Espérance » qui consiste à calculer lespérance conditionnelle de la log-vraisemblance des données complètes sous la loi des données non observées sachant les observations à litération courante, et une seconde étape de « Maximisation » dans laquelle on maximise lespérance conditionnelle obtenue lors de la première étape. Ces deux étapes seront détaillées plus loin.
Larticle fondateur est celui de Dempster, Laird, et Rubin 1977, dans lequel sont énoncés les principes généraux, et qui a donné son nom à lalgorithme, même si dautres auteurs ont développé avant eux des algorithmes similaires, mais dans des cas particuliers. Ainsi, la plus ancienne référence à un algorithme de type EM revient à Newcomb 1886 pour lestimation des paramètres dun modèle de mélange gaussien. Plus tard, Orchard et Woodbury 1972 déinissent le « missing information principle », et établissent le lien entre la vraisemblance complète et la vraisemblance incomplète. Sundberg 1974 propose également une étude détaillée de lalgorithme dans le cas particulier des modèles appartenant à la famille exponentielle. La convergence de lalgorithme sous des conditions générales de régularité a été démontrée par Dempster et al. 1977 ; Wu 1983 ; Boyles 1983. De nombreuses extensions ont été proposées depuis, comme par exemple lalgorithme ECM Meng et Rubin, 1993, pour le cas où létape de maximisation ne peu pas se résoudre explicitement, et est remplacée par une succession de maximisations conditionnelles. Wei et Tanner 1990 proposent également une extension de lalgorithme dans le cas où létape E nest pas explicite et est remplacée par une approximation de type Monte-Carlo. Le lecteur intéressé pourra se référer par exemple à louvrage de McLachlan et Krishnan 2007. Nous discuterons plus loin deux extensions de lalgorithme dans le cas où létape E nest pas explicite, à laide de lalgorithme MCMC-EM section 2.3 et de lalgorithme SAEM section 2.4.
La convergence de la séquence produite par lalgorithme EM vers le maximum de vraisemblance nest pas garantie, et en général, dans la plupart des applications, la convergence a lieu vers un point stationnaire de la vraisemblance, qui peut être un maximum local ou global, ou un point-selle. Sous certaines conditions supplémentaires de régularité, Wu 1983 a montré que lon peut sassurer de la convergence vers un maximum local. Cependant, ces conditions peuvent être difficiles à vériier dans la pratique, et lalgorithme peut alors se retrouver bloqué à un point stationnaire de la vraisemblance qui ne soit ni un maximum global, ni même un maximum local. Dans ces cas-là, une perturbation aléatoire du vecteur de paramètres peut permettre à lalgorithme de sen éloigner McLachlan et Krishnan 2007. Il sagit dun des avantages des versions stochastiques de lalgorithme EM.
Nous présentons ici la formulation générale de lalgorithme EM. En partant dune valeur initiale θ(0), litération k + 1 de lalgorithme consiste à réaliser successivement les deux étapes suivantes :
Étape E : on évalue lespérance conditionnelle de la log-vraisemblance des données complètes sous la distribution des données manquantes ou cachées sachant les observations, et sous lestimation courante de θk appelée Q ou Q-function en anglais :
| Q(θ; θk) = E (log f(x; θ) | y; θk) . | 2.6 |
|
Table des matières
Remerciements
Introduction
1 Développement et fonctionnement dune plante
1.1 Élements de morphogenèse végétale
1.2 Fonctionnement
2 Bref historique des modèles de croissance de plantes
3 Problématiques
4 Organisation du manuscrit et remarques préliminaires
1 Sélection de modèles pour la prévision
1 Modèles
1.1 Greenlab
1.1.1 Organogenèse
1.1.2 Allocation
1.2 LNAS
1.3 STICS
1.3.1 Production de biomasse
1.3.2 Croissance foliaire
1.3.3 Croissance racinaire
1.4 Pilote
1.5 CERES
1.6 Prise en compte des stress
2 Calibration
2.1 Données dapprentissage
2.2 Paramètres considérés comme ixes
2.3 Analyse de sensibilité
2.3.1 Principes généraux
2.3.2 Application
2.4 Sélection du nombre de paramètres
2.4.1 Méthode destimation
2.4.2 Critères de sélection
3 Prévision
3.1 Données test
3.2 Critères
3.2.1 Erreur quadratique moyenne de prédiction MSEP
3.2.2 Efficience de modélisation EF
3.2.3 Erreur relative de prédiction du rendement
3.2.4 Observations vs. prédictions
4 Résultats
4.1 Comparaison des différentes versions de STICS
4.2 Comparaison sur les données 2008
4.2.1 Masse totale
4.2.2 Masse racinaire
4.3 Comparaison sur les données 2011
4.3.1 Masse totale
4.3.2 Masse racinaire
5 Conclusion et perspectives
2 Généralités sur les modèles non linéaires mixtes
1 Formulation du modèle
2 Estimation dans le modèle non linéaire mixte
2.1 Les différentes approches
2.1.1 Méthodes basées sur lestimation des paramètres individuels
2.1.2 Méthodes basées sur une approximation de la vraisemblance
2.1.3 Méthodes « exactes »
2.1.4 Méthodes exactes basées sur lutilisation de lalgorithme EM
2.2 Lalgorithme EM
2.2.1 Le cas du modèle exponentiel
2.2.2 Intervalles de coniance
2.2.3 Convergence de lalgorithme
2.3 Lalgorithme MCMC-EM
2.3.1 Conditions dapplication du théorème ergodique
2.3.2 Algorithme de Metropolis-Hastings
2.3.3 Échantillonneur de Gibbs
2.3.4 Échantillonneur de Gibbs hybride
2.3.5 Taille de la chaîne et critère darrêt
2.3.6 Convergence de lalgorithme
2.4 Lalgorithme SAEM
2.4.1 Principe général
2.4.2 Convergence de lalgorithme
2.5 Estimation de la vraisemblance
3 Évaluation du modèle
3.1 Structure de covariance
3.2 Erreur de prédiction sur la distribution
3 Modélisation de la variabilité inter-plantes
1 Lorganogenèse chez la betterave
1.1 Formulation du modèle
1.2 Estimation sous Monolix
1.3 Données
1.4 Résultats
1.4.1 Population standard
1.4.2 Comparaison des doses dazote
1.5 Discussion
2 Le modèle Greenlab de population
2.1 Formulation du modèle
2.1.1 Variabilité intra-individuelle
2.1.2 Variabilité inter-individuelle
2.2 Estimation
2.2.1 Étape E
2.2.2 Étape M
2.2.3 Convergence de lalgorithme
2.2.4 Intervalles de coniance
2.3 Simulations
2.3.1 Algorithme MCMC-EM
2.3.2 Algorithme SAEM
2.4 Application sur données réelles
2.4.1 Données expérimentales
2.4.2 Résultats
2.5 Discussion
Discussion et perspectives
1 Principaux résultats et contributions
1.1 Sélection de modèles pour la prévision
1.2 Variabilité inter-individuelle
2 Perspectives
2.1 Sélection de modèles pour la prévision
2.2 Variabilité inter-individuelle
2.2.1 Modèle dorganogenèse
2.2.2 Modèle Greenlab de population
Annexes
A Paramètres des modèles du Chapitre 1
B Calcul de la matrice d’information de Fisher
Glossaire
Publications
Bibliographie
Télécharger le rapport complet