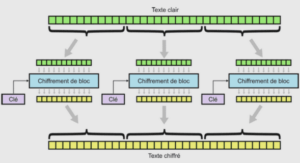
Le sang humain est un liquide physiologique en temps normal totalement stérile. Cependant des micro-organismes peuvent pénétrer dans le sang via une infection locale (lésion de la peau, ingestion d’aliments contaminés…) ou un traitement médical (chirurgie, pose d’un cathéter …). La répétition et/ou la prolongation de cette incursion de micro-organismes peut provoquer une infection sanguine, si la réponse du système immunitaire n’est pas suffisante. On recense environ 30 millions d’infections sanguines chaque année dans le monde. C’est une pathologie répandue mais également potentiellement fatale.
La prise en charge thérapeutique d’une infection sanguine repose sur la prescription d’antibiotiques, d’abord à large spectre, puis ciblés une fois le pathogène identifié. Cette stratégie occasionne donc un délai pendant lequel le patient est traité avec une antibiothérapie probabiliste et des molécules potentiellement peu ou pas adaptées à sa pathologie.
Un diagnostic rapide permet d’améliorer significativement les chances de survie du patient mais également de limiter un processus biologique bien connu et de plus en plus menaçant pour la santé mondiale : l’antibiorésistance. Responsable de plus d’un million de décès dans le monde en 2019, la recrudescence de germes bactériens résistants aux antibiotiques pourrait tuer 10 millions de personnes chaque année avant 2050, soit plus que le cancer.
Du fait de la très faible biomasse présente dans la prise de sang (quelques cellules à peine), il est nécessaire, pour dépister une contamination bactérienne (ou fongique), de placer les microorganismes à détecter dans des conditions favorables à leur prolifération. Ainsi, à partir d’une unique cellule, les microbes (bactéries ou champignons) se multiplieront jusqu’à atteindre des concentrations aisément détectables, par un automate ou un œil humain. La mise en culture d’une prise de sang pour en déceler les éventuels micro-organismes se nomme hémoculture. Il s’agit d’un test de stérilité, sans a priori sur la nature et le nombre des micro-organismes à détecter : la présence d’une seule cellule bactérienne ou fongique suffit à rendre le test positif.
Le XXème siècle a été une période dynamique dans le domaine des avancées technologiques pour l’hémoculture. Les procédés de détection, la sensibilité des capteurs/outils de détection et la fertilité des milieux de culture ont été améliorés. En effet, le développement d’une hémoculture rapide et à bas coût représente un enjeu majeur en terme de santé publique :
➔ Le taux de survie des patients serait amélioré grâce à la prescription plus rapide d’un traitement adapté ;
➔ L’apparition de « super-bactéries », résistantes à presque toutes les stratégies antimicrobiennes, pourrait potentiellement être retardée ;
➔ La technologie pourrait s’exporter dans les pays à faible revenu et à revenu intermédiaire, foyers de l’antibiorésistance et qui représentent une grande majorité des infections sanguines à l’échelle mondiale, afin de déployer une méthodologie accessible permettant de détecter et circonscrire les foyers infectieux.
Le suivi des hémocultures repose la plupart du temps sur une détection des métabolites émis par les micro-organismes en croissance. Du fait, de la difficulté à déceler optiquement ces métabolites dans le sang, même dilué, c’est dans une phase hétérogène adjacente, phase solide ou phase gazeuse, qu’est typiquement réalisée la détection de métabolites microbiens. En opposition à cette approche classique, cette thèse vise à étudier l’intérêt d’un suivi de l’hémoculture directement dans la phase liquide, complexe, par des mesures électrochimiques. En outre, alors que les méthodes conventionnelles reposent sur le suivi d’un seul paramètre, typiquement la génération de CO2 ou la pression globale, le présent manuscrit montre le bénéfice à réaliser un suivi multiparamétrique de l’hémoculture, de manière à extraire une « empreinte » électrochimique. Ainsi, l’instrumentation du flacon d’hémoculture par un réseau de capteurs électrochimiques assure non seulement la détection du micro-organisme responsable de l’infection mais rend également possible son identification, le tout sans requérir la manipulation du sang infecté par une intervention humaine ni ajout de réactifs. De par l’approche applicative du sujet, les problématiques d’industrialisation et d’utilisation (notamment en milieux isolés) ont été prises en compte.
Etat de l’art sur le test d’hémoculture et le contexte de la thèse
Le test d’hémoculture
Le sang est un liquide physiologique composé de plasma et de cellules appelées éléments figurés du sang. Le sang sain, à l’instar de l’urine et du liquide cérébrospinal, est un liquide stérile, c’est-à-dire que sa mise en culture ne produit aucune croissance de micro-organismes (bactéries ou levures). Cette caractéristique permet de détecter facilement un sang infecté par un test de stérilité, l’hémoculture. Son principe est simple : un échantillon de sang est prélevé sur le patient, puis mis en culture dans un milieu nutritif favorisant la croissance de microbes, à une température permettant cette croissance (située entre 34 et 37°C). Cette culture est nécessaire pour augmenter la biomasse des microorganismes, biomasse très faible dans le corps du patient (parfois 10⁻² cfu* /mL) malgré des symptômes potentiellement très graves . En effet, en médiane chez l’adulte, une concentration de 1 cfu par millilitre de sang est retrouvée pour une infection sanguine. On appelle bactériémie une infection sanguine causée par une bactérie, alors qu’une fongémie sera causée par une levure dans le sang.
Contexte
Les bactériémies dans le monde
Les infections sanguines sont un problème de santé mondial : on estime à plus de 30 millions le nombre d’épisodes de bactériémies par an dans le monde, entraînant 6 millions de décès . Non traitée, une bactériémie peut conduire à une septicémie (ou sepsis) puis à un choc septique. Le taux de mortalité d’un sepsis est très élevé, entre 25 et 50 %. Selon l’Organisation mondiale de la santé, les septicémies sont responsables de près de 20 % de tous les décès dans le monde . La majorité des infections sanguines sont mono-microbiennes, ce qui facilite l’identification du pathogène responsable, mais les infections polymicrobiennes, plus complexes à traiter, représentent jusqu’à 30 % des bactériémies chez certaines populations. Un diagnostic complet est souvent beaucoup plus long que la durée pendant laquelle la décision thérapeutique doit être prise par le praticien. C’est cette différence qui conditionne l’utilisation d’antibiotiques à large spectre dans le but de gagner du temps jusqu’à la détermination du traitement le plus adapté.
Malheureusement, dans 20 % des cas de sepsis, le traitement initial est inapproprié, ce qui augmente fortement le taux de mortalité pour ces patients . Cette étude de Kumar et al. rapporte même une multiplication du taux de mortalité par un facteur 5 chez les patients avec une mauvaise stratégie antimicrobienne. En plus de cette conséquence, potentiellement létale, à court terme, l’utilisation d’une antibiothérapie peu ou pas adaptée favorise la sélection de souches bactériennes antibiorésistantes.
En 2019, plus de la moitié des souches de Staphylococcus aureus isolées étaient résistantes à la méthicilline dans de très nombreux pays telles que la Chine, l’Inde mais également les Etats-Unis . L’antibiorésistance est donc une réalité proche géographiquement.
Il existe des organismes nationaux et internationaux, tels que l’EUCAST (European Committee on Antimicrobial Susceptibility Testing), spécialisés dans la surveillance de ces souches antibiorésistantes ainsi que dans les bonnes pratiques hospitalières pour limiter leur prolifération . Pourtant, force est de constater que la résistance aux antibiotiques progresse toujours, au point d’être qualifiée par l’OMS de « l’une des plus grandes menaces pour la santé mondiale, la sécurité alimentaire et le développement aujourd’hui ». Une étude récente fait état de plus de 1,27 million de décès directement causés par la résistance aux antimicrobiens en 2019 ; ce nombre pourrait atteindre 10 millions chaque année d’ici 2050.
|
Table des matières
INTRODUCTION GENERALE
CHAPITRE 1 ETAT DE L’ART SUR LE TEST D’HEMOCULTURE ET LE CONTEXTE DE LA THESE
1. LE TEST D’HEMOCULTURE
1.1. Contexte
1.1.1. Les bactériémies dans le monde
1.1.2. Prise en charge des bactériémies
1.2. Les différentes méthodes de détection pour le test d’hémoculture
1.2.1. Les débuts de la détection des infections sanguines
1.2.2. Les détecteurs automatisés de dioxyde de carbone
1.2.3. Les automates utilisant l’électrochimie
1.3. L’identification bactérienne
1.3.1. Identification après culture
1.3.1.1. Méthodes métaboliques
1.3.1.2. Spectrométrie de masse
1.3.1.3. Méthodes génomiques
1.3.1.4. Méthodes immunologiques
1.3.2. Identification pendant la culture
1.3.3. Les technologies de rupture
2. APPORT DE L’ELECTROCHIMIE POUR LA DETECTION ET L’IDENTIFICATION BACTERIENNE
2.1. Détection de métabolites bactériens
2.1.1. Mesure directe
2.1.2. Mesure indirecte
2.2. Fixation d’un micro-organisme sur une sonde
2.2.1. Utilisation d’un anticorps
2.2.2. Utilisation d’une sonde ADN/ARN
2.2.3. Utilisation de bactériophages
2.3. Méthodes annexes
2.4. Conclusions sur l’intérêt de l’électrochimie en microbiologie
3. L’APPROCHE DE LA THESE : L’ANALYSE ELECTROCHIMIQUE SANS A PRIORI
3.1. Problématiques des hémocultures actuelles
3.2. Le flacon instrumenté par des capteurs électrochimiques
CHAPITRE 2 MATERIELS ET METHODES
1. RAPPELS THEORIQUES
1.1. Bactéries et métabolisme
1.1.1. Définitions
1.1.1.1. Les biofilms bactériens
1.1.1.2. Le Gram des bactéries
1.1.2. Métabolisme bactérien
1.2. Mesure du pH
1.2.1. Indicateur colorimétrique
1.2.2. Le pH-mètre
1.2.3. Les encres conductrices
2. CHOIX DE LA METHODE DE MESURE
2.1. Description de la potentiométrie
2.2. Description de la spectroscopie d’impédance
3. FABRICATION DU BANC DE MESURE
3.1. Cahier des charges du dispositif de mesure
3.2. Conception des électrodes
3.2.1. Electrode de travail
3.2.1.1. L’encre polyaniline
3.2.1.2. L’encre oxyde d’iridium
3.2.1.3. Les encres carbonées sans oxyde métallique
3.2.2. Electrode de référence
3.3. Instrumentation du flacon
3.4. Protocole d’ensemencement et de mesure
3.5. Utilisation de différentes matrices
3.6. Choix des micro-organismes à étudier en priorité
CHAPITRE 3 DETECTION ET IDENTIFICATION DES BACTERIES PENDANT LEUR CROISSANCE
1. SUIVI DU PH PENDANT UNE HEMOCULTURE
1.1. Mesures électrochimiques avec la polyaniline
1.2. Mesures électrochimiques avec l’oxyde d’iridium
1.3. Corrélation entre pH, biomasse et potentiels
1.3.1. Protocole de l’expérience réalisée
1.3.2. Résultats et discussion
1.3.2.1. Calcul des limites de détection
1.3.2.2. Influence du pH du milieu sur le potentiel des électrodes
1.3.3. Conclusion
1.4. Utilisation de matériaux non sensibles au pH
1.4.1. Mesures électrochimiques avec une encre carbone
1.4.2. Mesures électrochimiques d’une encre « bleu de Prusse »
1.5. Hypothèses de travail
2. SUIVI ET DETECTION D’UNE HEMOCULTURE POSITIVE
2.1. Evolution des potentiels en l’absence de bactérie
2.2. Utilisation de la dérivée du potentiel
2.3. Etude de la réponse de la polyaniline pour une croissance d’E. coli
2.3.1. Influence de l’inoculum
2.3.2. Détection de souches différentes
3. BASE DE DONNEES SUR PLUSIEURS ESPECES BACTERIENNES
3.1. Les bactéries à Gram négatif
3.1.1. Les entérobactéries
3.1.2. Les autres bactéries à Gram négatif
3.2. Les bactéries à Gram positif
3.2.1. Les staphylocoques
3.2.2. Les autres coques à Gram positif
3.3. Détection de fongémies
4. LE CALCUL DU TEMPS DE DETECTION
5. IDENTIFICATION PAR APPRENTISSAGE MACHINE
5.1. Traitement des données
5.1.1. Choix d’un descripteur adapté
5.1.2. Choix d’un classifieur adapté
5.2. Performances d’identification
5.2.1. Identifier le Gram d’une bactérie
5.2.2. Identifier le genre d’une bactérie
5.2.3. Algorithmes « croisés »
5.3. Contribution des différents matériaux d’électrodes pour l’identification
6. CONCLUSION
CHAPITRE 4 ETUDE DU PHENOMENE OBSERVE PAR POTENTIOMETRIE
1. INFLUENCE DES PARAMETRES EXPERIMENTAUX SUR LE SIGNAL MESURE
1.1. Influence du milieu de culture
1.2. Influence de l’agitation et de la température
1.2.1. Abaissement de la température de culture
1.2.2. Suppression de l’agitation
1.2.3. Mesure dans le surnageant
1.3. Influence de la surface d’électrode
2. HYPOTHESE DE LA DETECTION D’UN BIOFILM BACTERIEN
2.1. Mesures potentiométriques avec filtration par diffusion
2.1.1. Objectifs de l’expérience
2.1.2. Résultats
2.1.3. Hypothèse concernant le fonctionnement de notre détection
2.2. Etude par spectroscopie d’impédance
2.2.1. Objectifs de l’expérience
2.2.2. Résultats et discussion
2.2.2.1. Impact de la mesure EIS sur la détection
2.2.2.2. Evolution de l’impédance pendant la croissance : le cas de la polyaniline
2.2.2.3. Etude de l’impédance à 0,1 Hz
2.3. Observations par microscopie numérique
3. CONCLUSION
CONCLUSION GENERALE