Code barre ADN et séquençage 454
MATERIEL ET METHODES
Matériel biologique
Site expérimental
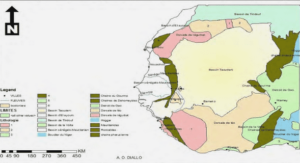
La parcelle expérimentale du CIRAD est située sur le site de Rivère Lezarde (60° 59′ 59 » W; 14° 39′ 45 » N) (Annexe 1(A)). Elle est constituée d’un bloc « sol Nu » (sol N) et d’un bloc « sol enherbé »(sol E). Le sol N est composé de deux rangées de bananiers et d’une interrangée. Le sol E regroupe quatre rangées de bananiers et les trois inter-rangées. Les deux blocks sont séparés par une rangée de bananier sur sol enherbé, cette séparation est exclue de l’expérimentation. Le bloc nu a été désherbé régulièrement avec du glyphosate. La végétation présente sur le sol enherbé se compose majoritairement de Brachiaria decumbens (Poaceae).
Échantillonnage et identification
Flore
Nous avons échantillonné la flore à l’intérieur et en bordure de la parcelle expérimentale de façon exhaustive, dans la mesure du possible (voir plan Annexe 1(B)).
Nous avons prélevés 5 spécimens par taxons visuellement différencié (cas d’espèces très peu abondantes sur la parcelle, représentées par un ou deux individus). Chaque espèce est au moins représentée par un spécimen. Sur chaque spécimen, nous avons prélevés au moins 100mg de feuille, tissu qui contient le plus de chloroplastes (pour l’extraction de l’ADN chloroplastique).
Les premières déterminations systématiques ont été réalisées à partir des échantillons bruts et de support photos (photos prises sur le terrain). Ces déterminations ont été réalisées à l’aide d’une clé de détermination (Fournet, 2006) et de manuelles de références (Jacques Fournet et Hammerton, 1991). Par la suite, les séquences d’ADN obtenues pour ces échantillons ont été comparées et identifiées sur Genebank®.
Faune
Les échantillonnages ont été effectués de février à Avril 2011.Les déterminations systématiques des organismes échantillonnés ont été effectuées sous une loupe binoculaire. La reconnaissance a été faite au niveau de la famille avec une clé de détermination (Delvare et Aberlec, 1989) et avec l’aide d’un entomologiste de l’équipe, M Philippe Ryckewaert.)
Nous avons utilisés trois méthodes de piégeage:
– Pièges de Barber (ou piège fosse). Il s’agit d’un contenant enterré dans le sol, fermé par un couvercle (protection). Les parois sont lisses et une collerette est en plus placée au sommet du piège; les insectes qui y tombent ne peuvent plus ressortir. Le sol a été creusé et les pièges ont été disposés le long de chaque rangée et inter-rangée, à intervalle régulier sur les deux blocks à raison de 5 pièges par rangée (quatre pièges pour la dernière rangée sol E, plus petite). Dans certains pièges, des capsules de la phéromone d’aggrégation (Cosmolure®,Costa Rica), la sordidine, sont ajoutées afin d’augmenter quantitativement la capture de charançons. (Ce biais dans l’abondance des charançons explique pourquoi leur abondance n’a pas été mesurée).
– « Pseudo tronc ». Des sections de pseudo tronc, cinq par rangée, ont été placées dans toutes les rangées de bananiers, sur les deux sols, entre les pièges « fosse ». Ils ont été changés régulièrement lorsqu’ils étaient desséchés; soit environ tous les quinze jours.
– Aspirateur. Pour l’épifaune et les organismes volants sur les deux sols.
Afin de limiter la dégradation de l’ADN, chaque individu capturé a été placé dans un tube rempli d’alcool (70°), et directement disposé dans une glacière réfrigérée sur le terrain, avant d’être stocké au congélateur (- 20°C) au laboratoire.
Extraction ADN
Échantillons végétaux
Parmi les échantillons récoltés, quatre individus par espèces (quatre répétitions) sont utilisés pour les extractions d’ADN (le dernier est conservé au laboratoire), à l’exception des espèces faiblement représentées sur le site d’échantillonnage pour lesquelles nous avons extrait l4ADN des individus disponibles. Ces extraits d’ADN ont permis de constituer une librairie de référence du gène trnL pour toutes les espèces végétales présentes à l’intérieur et en ordure de la parcelle expérimentale. Les échantillons ont été séchés pendant 48H dans un lyophilisateur puis réduit en poudre avec deux billes en métal d’un diamètre de 5mm placées dans le tube, et vortexés pendant une dizaine de secondes. Pour limiter les contaminations croisées, les billes sont nettoyées avec une solution qui dégrade les acides nucléiques (DNA Away ®). La poudre est conservée dans un nouveau tube stérile au réfrigérateur 24H avant l’extraction d’ADN.L’ADN total a été extrait à partir de 20mg de poudre avec le kit d’extraction « DNeasy Mini Plant Kit » (Qiagen®, Venlo, Pays-Bas), en suivant le protocole standard recommandé. Chaque échantillon d’ADN extrait est conservé dans 100µL de tampon d’élution.
|
Table des matières
I.INTRODUCTION
II. OBJECTIFS
III. MATERIEL ET METHODES
1. Matériel biologique
1.1. Site expérimental 5
1.2. Échantillonnage et identification
1.2.1. Flore
1.2.2. Faune
1.3. Extraction ADN
1.3.1. Échantillons végétaux
1.3.2. Contenus stomacaux
2. Analyses isotopiques
2.1. Principe
2.2. Traitements des échantillons
3. Marqueurs moléculaires
3.1. Amplification par PCR
3.1.1. Mise au point du protocole expérimental
3.1.2 Amorces spécifiques
3.2. Code barre ADN et séquençage 454
3.2.1. Constitution librairie
3.2.2. Contenus stomacaux
4. Analyses statistiques
4.1. Analyses isotopiques
4.2. PCR 16
4.3. Code barre ADN
IV. RESULTATS
1. Échantillonnages
1.1. Faune
1.2. Flore
2. Analyses Isotopiques
3. Marqueurs moléculaires
3.1. Mise au point du protocole expérimental
3.3. Amplification PCR avec amorces spécifiques
3.4. Séquençage 454
V. DISCUSSION
IV. CONCLUSION
Références Bibliographiques
Liste des abréviations
Annexes
![]() Télécharger le rapport complet
Télécharger le rapport complet